Comment le labo a utilisé HAL
Ce post est un recueil d’expériences menées au sein du laboratoire d’informatique du LIMOS, il explique les raisons pour lesquelles le labo s’est intéressé à HAL et quels moyens il a mis en œuvre pour parvenir à intégrer cette plateforme dans son fonctionnement quotidien.


Sommaire
Le choix de HAL
Récupération des publis
Base de données
Insertion dans la base
Ranking des publications
Qualité et exhaustivité des données
Un travail d'équipe
Résultats
Le choix de HAL s’est fait pour plusieurs raisons.
En premier lieu, il s’agit d’une recommandation du CNRS.
La recherche publique française souhaitait voir s’affaiblir la pleine puissance des éditeurs privés (Elsevier, Springer, …) ; en plus de la promotion d’outils de l’OpenScience, elle a émis une série de textes juridiques visant à protéger les travaux des chercheurs pour que ceux-ci gardent la propriété de leurs publications.
Ainsi, il est possible de déposer sur HAL, sans que l’éditeur ne puisse l’empêcher, une version PrePrint, une version PostPrint et même une version éditeur dans certains cas (PDF en accès libre, clause d’embargo de durée définie, ...)
Parallèlement, le LIMOS souhaitait développer un système d’information afin de mieux maîtriser ses données. Il m’a été demandé d’en construire un qui puisse à la fois être une vitrine (un site web de présentation du labo) et une base de données afin de regrouper et recouper les données des fichiers Excel des différents administratifs du labo.
Le choix des outils s’est porté sur MySQL pour sa simplicité et sur Django (framework python) pour sa facilité à construire un backend.
La question s’est rapidement posée de savoir quoi présenter en terme de production scientifique. Le labo étant divisé en axes et thèmes de recherche, la direction du labo souhaitait savoir quel thème publiait quoi et voulait aussi avoir un état exhaustif et complet des publications réalisées au LIMOS.
Nous avions en équipe déjà réfléchi à la plateforme HAL mais ne savions pas comment l’utiliser efficacement. Plusieurs chercheurs du LIMOS avaient déjà publié sur HAL et une base de départ existait mais nous souhaitions y intégrer la notion de thèmes de recherche.
Après une présentation de certaines utilisations de HAL par le biais du réseau ARAMIS, nous avons convenu du process suivant : tous les chercheurs devraient dorénavant déposer leurs travaux sur HAL, nous exporterions ensuite l’ensemble des publications du LIMOS dans notre base de données et affecterions à chaque publication un ou plusieurs thèmes de recherche.
En fait, il existe un champ assez proche de cette notion d’équipe (ou de thème de recherche) dans HAL mais comme ce n'est pas forcément le chercheur en question qui va déposer mais un co-auteur ne connaissant pas forcément le nom de l’équipe du chercheur, comme ces équipes peuvent changer dans le labo sans répercussion possible dans HAL, cela ne semblait pas une bonne idée de renseigner dans HAL le nom de l’équipe du chercheur pour chaque publication.
Comme j'étais en même temps chargé de travailler sur le SI (système d'information) du LIMOS, j'ai donc récupéré toutes les publications de la collection du LIMOS en récupérant des champs bien spécifiques (les noms des auteurs et leur idHal, le titre, le type de document, la date mais aussi le titre du journal de parution ou de la conférence)
L’idée étant d’associer à chaque publi une ou plusieurs personnes si elles étaient co-auteur de la publi et un ou plusieurs thèmes en fonction du thème associé à ces personnes
Je me suis alors intéressé à l’API de HAL permettant de récupérer du contenu à partir du portail de HAL
https://api.archives-ouvertes.fr/docs/search
Le langage de requête de HAL est basé sur le langage de requête d'Apache Solr.
Les publications d’un labo étant regroupées dans une collection, il est simple de récupérer toutes les publications affiliées à un labo en ajoutant son nom au point d’entrée de l’API :
https://api.archives-ouvertes.fr/search/LIMOS
Ici, j’ai choisi de filtrer ma requête :
récupération de toutes les données → q=*:*
concaténation des différents filtres → &
quantité de données recherchées → start=0&rows=5000
tri par date de publication décroissante → sort=producedDate_s%20desc
Puis, j’ai récupéré seulement certains champs des nombreux champs qu’il est possible d’extraire :
https://api.archives-ouvertes.fr/docs/search/?schema=fields#fields
On sélectionne le paramètre indiquant que l’on veut choisir des champs → fl=
Puis on ajoute successivement les champs séparés par une virgule
- uri_s : URL de la publication
- halId_s : identifiant HAL de la publication
- authFullName_s : noms et prénoms des co-auteurs
- authIdHal_s : identifiants HAL des coauteurs
- title_s : titre de la publication
- docType_s : type du document (article, conférence, ouvrage, thèse, ...)
- keyword_s : liste des mots clés renseignés
- producedDate_s : date de publication
- domain_s : domaines associés à la publi (sélection fermée)
- journalTitle_s : titre du journal dans lequel est paru l’article
- conferenceTitle_s : tire de la conférence à laquelle l’auteur a participé
On arrive à la requête finale renvoyant du json (possibilité de spécifier l’export)
https://api.archives-ouvertes.fr/search/LIMOS/?q=*:*&start=0&rows=5000&sort=producedDate_s%20desc&fl=uri_s,halId_s,authFullName_s,authIdHal_s,title_s,docType_s,keyword_s,producedDate_s,domain_s,journalTitle_s,conferenceTitle_s
Concernant la construction du SI, j’ai utilisé une base de données MySql
Côté base, j’avais récupéré dans des tables les identités de tous les membres du labo avec leur affectation par thème de recherche (un ou plusieurs par chercheur)
Ceux-ci, comme les doctorants, peuvent modifier certaines informations de leur identité sur la base. Je leur ai permis entre autre de renseigner un champ "idHAL".
Les tables « Person », « Publication »et « Theme » sont reliées entre elles par des clés étrangères et comme une personne peut avoir ‘n’ publications, une publication appartenir à ‘n’ personnes, … , des tables relationnelle notées mtm_ relient ces trois tables.
Le schéma ci-dessous représente les tables de la partie de la base correspondante :
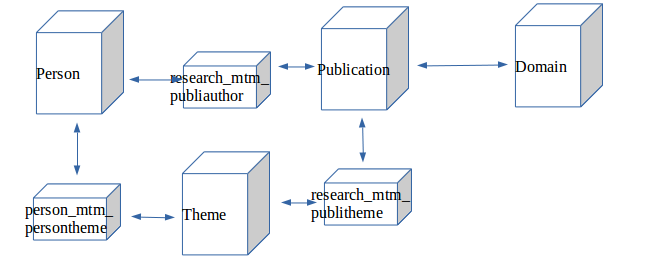
Ainsi, pour enregistrer une publication dans la base, on utilise la table ‘publication’.
Pour la relier à une personne, on écrit dans la table ‘research_mtm_publiauthor’ en ajoutant l’id de la publi et l’id de la personne.
Pour la relier à un thème, on écrit dans la table ‘research_mtm_publitheme’ en ajoutant l’id de la publi et l’id du thème.
Pour la relier à un domaine, on enregistre dans la table ‘Domain’ l’id de la publi et les différents domaines.
J’ai réalisé un script python permettant de remplir la table publication à partir de l’export de HAL
Étant donné que HAL va corriger régulièrement sa base, merger des dépôts, en retirer, ... , toutes les publications sont supprimées de la base toutes les nuits et recréées immédiatement.
Ce premier script script_populate_publication_renew.py est visible sur le dépôt Gitlab :
https://gitlab.limos.fr/basdorea/hal-limos
Ce script se déroule en 5 étapes
- 1. Récupération par une requête sur l'API HAL de toutes les publis du LIMOS (avec des champs spécifiques). Le résultat de la requête en JSON est parsé ; les champs uniques (URL, title, …) sont récupérés directement et les champs multiples (authors, domains, …) enregistrés dans des listes. Toutes ces informations liées à une publication sont ajoutées dans une liste 'publilist' (lignes 52 – 289)
Pour les domaines, un dictionnaire de correspondances est fait pour une meilleure compréhension - 2. Suppression dans la base du contenu des tables mtm_publiauthor, mtm_publitheme, publication et domainpublis. (lignes 293 – 303) L’id max de la table publication est 0 -> cptPubli=0
- 3. Récupération par une requête de la base des informations sur les personnes, notamment les champs id, authHalId, first_name, last_name qui sont enregistrés dans une liste ‘listPerson‘
Récupération dans le même temps de l’id du thème principal de la personne (lignes 309 – 333) - 4. La base étant vide, on insère toutes les publis.
- On commence par insérer les domaines (table domain), puis la publication à partir des éléments de la liste publislist.
- Pour chaque insertion de publi, on met en parallèle les listes publislist et listPersons ; si un des éléments idHal de publislist est égal à un des éléments idHal de la liste listPersons , c’est que la personne est un des co-auteur et on fait une insertion dans la table research_mtm_publiauthor.
- Si la personne n’a pas de idHal, on compare la forme auteur de HAL (prénom + nom) avec le prénom et le nom de la personne (*).
- Dans les deux cas, dès qu’un matching est fait avec une personne, on associe le thème principal (le premier) de la personne avec la publication en faisant une insertion dans la table research_mtm_publitheme (lignes 354 – 446) - 5. Enregistrement des résultats et problèmes éventuels dans des logs (lignes 450 – 469)
(*) : cette solution est peu fiable car sur HAL, un chercheur peut avoir des formes auteurs différentes (juste la première lettre du prénom, oubli des accents, …), la meilleure solution est que chaque chercheur ait un idHAL
Une fois fait, il est possible de filtrer facilement ces publications. Ainsi, j’ai pu proposer dans le site web un filtre par thème, par auteur ou par type de document et proposer à chaque fois un lien vers le document dans HAL. Les PDF des articles ne sont pas dans le SI mais sont accessibles en deux clics. Le filtre par type de document peut s’avérer très intéressant pour obtenir le nombre de thèses, d’HDR, ...
J’ai aussi pu présenter en page d’accueil une sélection de 10 articles parmi les 20 derniers publiés ou encore présenter pour chaque thème les publication lui étant associées.
Enfin, une fonction d’export de tables sous Excel permet de récupérer tous les champs de toutes les publis de la table dans un tableur afin de générer différents indicateurs sous forme de graphiques.
Lorsque je discutais avec des chercheurs de l’intérêt de déposer sur HAL, une des critiques les plus récurrentes était le manque d’information sur la qualité de la publication.
Le fait d’avoir les données en base a pris, ici, toute son importance.
En voyant que des organismes de ranking les plus courants dans le domaine de l’informatique, ScimagoJR et Core, proposaient des exports, j’ai pu croiser ces données avec celles que j’avais en base.
Scimago note uniquement les journaux dans de nombreux domaines scientifiques (notation de Q1 à Q4) et Core note les journaux et conférences uniquement dans le domaine informatique (notation de A* à C).
J’ai rajouté deux champs dans la table Publication pour chacune des notes et récupéré les CSV de Scimago et Core ; puis ai croisé les noms des journaux/conférences des CSV avec les noms des journaux et conférences rentrées en base.
Les premiers résultats étaient mitigés, une proportion honnête des articles avaient une note mais les conférences étaient très peu notées.
Je me suis aperçu que si les chercheurs remplissent exactement le titre du journal, ce n'est pas le cas pour la conférence. Parfois, l’année est rajoutée au titre de la conférence ou le chercheur indique seulement l’acronyme de la conférence, …
J’ai donc retouché les données en formatant la casse, supprimant les espaces latéraux, recherchant le nom exact du journal/conf dans le contenu du nom en base (au lieu d’un matching strict). Enfin, j’ai récupéré les acronymes des conférences et pour chaque conférence de Core, si le nom n’était pas retrouvé, je cherchais l’acronyme.
Malgré quelques faux positifs, cette méthode a été retenue car traitant le plus grand nombre de données.
Finalement, j’ai automatisé la création des fichiers CSV et rajouté deux scripts au premier, un pour créer des CSV ad hoc à partir de Scimago et Core et un autre pour comparer les données.
Ainsi, chaque nuit, les trois scripts suivants sont lancés dans cet ordre d'apparition :
script_populate_publications_renew.py -> supprime et renouvelle les publis à partir de la collection HAL du LIMOS
script_create_csv_scimago_core.py -> récupère les bases de données de scimago et Core et crée des fichiers CSV ad hoc
script_note_publications.py -> compare les infos entre les CSV et les journaux et conférences des publis enregistrées en base et leur attribue une note quand c'est possible
Qualité et exhaustivité des données
Le contenu sur la collection HAL du LIMOS dépend exclusivement des chercheurs et certains ont parfois rentré des informations succinctes quand d’autres n’ayant jamais déposé se sont retrouvés avec des centaines de publications à déposer.
Ainsi, la qualité des dépôts peut perturber le ranking et l’exhaustivité est dure à obtenir.
HAL propose une API particulière : SWORD, qui permet d'importer automatiquement et massivement dans HAL.
Le principe étant d'envoyer des XML contenant des données sur les publis sur une URL de SWORD.
Dans l'idée, je souhaitais pouvoir importer dans HAL des publications à partir d'une liste, après vérification que la publi n'existe pas déjà.
Cela résoudrait des problèmes pour les chercheurs ayant beaucoup publié mais peu déposé dans HAL.
Au départ, j'ai cherché un format de présentation des publications le plus générique possible à partir duquel je pourrais récupérer les données.
Le format Bibtex m'a paru une bonne idée mais de nombreux chercheurs n'utilisent pas ce format et certains ont seulement une liste de publis sous Word.
Je suis parti sur le format de publications demandé par l'HCERES étant donné que les chercheurs connaissent forcément ce format (ils ont fourni une liste à cet organisme dans les 5 dernières années) et que les administratifs du labo peuvent aussi me fournir de telles données.
Le souci étant que si des éléments de ce format se retrouvent bien à la bonne place, il existe des petites nuances dues à des formatages particuliers de Word ou LibreOffice Writer (« au lieu de ", noms et prénoms des auteurs formatés de façon différente, ...)
Ainsi un premier travail consiste à modifier cette liste avec un éditeur de texte pour le reformater correctement à partir de "Rechercher/Remplacer"
(Il est sans doute possible d'automatiser certaines tâches de formatage mais cette étape manuelle est nécessaire car un même chercheur indiquera dans sa liste un auteur avec le nom suivi du prénom puis, un peu plus loin, le prénom suivi du nom)
Puis j'ai écrit un script script_list_to_hal.py (python 2.7) permettant de lire cette liste et de récupérer les données nécessaires à la création de la publi.
Ce script génère pour chaque ligne de la liste (pour chaque publi) un fichier XML différant un peu en fonction du type de publi, puis génère à la fin un fichier CSV regroupant les données pour une meilleure lisibilité.
En spécifiant quelques variables en début de script, on autorise ou non le dépôt dans la base HAL de préproduction ou dans la base de production.
HAL demandant pour chaque conférence un nom de pays et un code correspondant au pays, le script va lire un fichier CSV de correspondance entre un pays et un code.
Cela implique d'avoir un nom de pays correctement écrit et se retrouvant dans cette liste.
Une fois la liste correctement formatée, le script se lance sans erreur et génère les XML. Lorsque ceux-ci sont importés dans une des bases HAL, la réponse HTTP est analysée pour savoir quel dépôt s'est bien ou mal passé.
Pour les premiers tests que j'ai pu faire, le taux d'import est de 80% à 90%.
Pour les XML n'étant pas passé, il est possible de les importer manuellement un par un. Un message d'erreur plus complet est renvoyé ce qui permet de corriger le XML afin de l'importer correctement.
L'intérêt de cette API est aussi de pouvoir déposer en donnant les droits de propriété (option on-behalf) à une personne bien précise sans avoir besoin de son mot de passe
Les fichiers (script et exemple de liste) se trouvent ici : https://gitlab.limos.fr/basdorea/hal-limos/tree/master/sword
Cette API permet aussi à partir d'un export de DBLP d'un chercheur au format Bibtex (càd des informations fiables et justes) de faire des dépôts sur HAL.
Un deuxième script script_dblp_to_hal permet de créer des dépôts à partir d'un fichier .bib ; le gros avantage étant qu'il n'est pas nécessaire de retoucher ce fichier bibtex mais l'inconvénient étant que seule une partie des publications est présente dans DBLP.
Dans l'idéal, il devrait être possible de faire juste certaines modifications de dépots (un PUT au lieu d'un POST) notemment concernant les noms des journaux et conférences, ce qui permettrait d'améliorer le ranking des articles et conférences en améliorant le matching.
Si le labo a pu intégré cette plateforme dans son quotidien , c’est grâce au concours de plusieurs personnes intervenues à différents moments.
Vincent Mazenod, IE au Limos, a participé aux prises de décisions stratégiques et aux différentes rencontres entre les acteurs et m’a beaucoup aidé et guidé pour la construction du SI.
Le réseau ARAMIS nous a permis de voir différents cas d’utilisation de HAL.
Pascal Lafourcade, chercheur, a fait le bêta-testeur sur HAL et m’a bien orienté sur les besoins des chercheurs tout en prodiguant des conseils avisés (« Récupère les journaux et conférences de HAL, ça pourra être utile. »)
Jessica Leyrit, administratrice HAL à la bibliothèque de l’UCA a répondu à nos questions sur HAL, a corrigé plusieurs problèmes dans la collection et a tenu des sessions de formation et d’aide pratique au sein du labo.
Le CCSD qui a développé HAL a pu nous aider pour des questions très techniques.
La direction a travaillé pour l’intégration de HAL en engageant formellement le labo à utiliser cette plateforme et en poussant les chercheurs à déposer dessus.
Au final, voilà ce que l'on peut avoir (seulement pour les membres connectés pour l'instant) :
Dans l'exemple ci dessous, les résultats pour un chercheur qui a bien fait attention à ses dépôts sur HAL

En dehors de l’aspect vitrine, chaque chercheur qui dépose correctement peut contrôler la qualité et l’exhaustivité de ses publications et la direction peut avoir des indicateurs sur la production scientifique pour chaque thème de recherche, dans le temps, par chercheur, …
L’HCERES auditant le labo cette année, ces outils pourront servir à rédiger le rapport efficacement.
Bastien DOREAU - publié le 4 Mars 2019

